基因是生物的遗传密码,通过基因编辑对生物进行特征改造或疾病治疗可以说是直击根本。工欲善其事,必先利其器,想要在基因水平上进行操作,必须要有“称手”的工具。在过去的数十年间,科学家们不断从自然界中“取材”,先后开发出了Cre-lox重组技术、锌指核酸内切酶(ZFN)技术、转录激活样效应核酸酶(TALENs)技术、CRISPR/Cas系统等工具。然而,现有的这些工具依然存在不够精准、编辑范围有限、难以递送等局限性,因此,科学界从未停止开发新基因编辑工具的脚步。

继3月30日,基因编辑先驱张锋领导的团队在Nature上发表论文,报道了一种可递送任何蛋白至任何细胞的系统后(我们曾在《张锋团队最新研究:可将蛋白递送至任何指定人类细胞,或打破基因治疗困局》一文中有过解读),短短一周后的4月6日,张锋团队又在Science发表最新论文“Structure of the R2 non-LTR retrotransposon initiating target-primed reverse transcription”,评估了家蚕(Bombyx mori)R2非LTR逆转录转座子作为新型基因编辑工具的潜力。

图1 研究成果(图源:[1])
所谓逆转录转座子,是基因组中一段有能力通过各种手段产生自己的“副本”并插入至其他位置的基因序列。其大致过程为,逆转录转座子先被转录成RNA并翻译出相应的蛋白,随后,生成的这些RNA和蛋白组成复合物,在基因组中找到合适的位置,进行逆转录和插入。
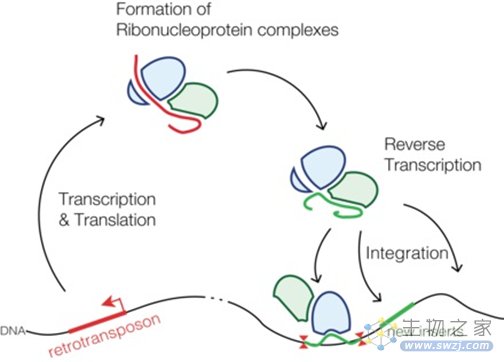
图2 逆转录转座子的生命周期示意图(图源:维基百科)
而非长末端重复序列(non-LTR)逆转录转座子,顾名思义,则指的是这类逆转录转座子的两端没有长链的重复DNA序列。在人类身上,非LTR逆转录转座子构成了基因组的17%。

图3 LTR和非LTR逆转录转座子的区别(图源:[2])
非LTR逆转录转座子又可进一步分成两类:LINEs(long interspersed nuclear elements)和SINEs(short interspersed nuclear element)。前者能够编码出“复制粘贴”所需的必要蛋白,而后者则做不到“自给自足”。
和所有的非LTR逆转录转座子一样,这次的主角,来自家蚕的R2元件(R2Bm),可以编码出具有结合DNA、切割DNA和逆转录功能的蛋白。负责切割的限制性核酸内切酶在“粘贴位置”,即目标DNA上切开口子,然后逆转录酶从暴露的3'端启动R2 RNA的逆转录,使得R2元件的新拷贝得以“安家落户”。这一过程被称为靶向启动逆转录(target-primed reverse transcription,TPRT)。
过去的研究表明,R2元件只会特异性地识别28S rRNA基因并插入到其内含子区域中。这种插入会导致28S rRNA基因的表达受到影响,或导致28S rRNA基因的突变和进化,从而影响到蚕的生长、发育、遗传多样性及进化。
然而,关于R2元件是如何识别28S rRNA基因,以及其编码出的蛋白如何在切割目标DNA后完成逆转录,这两个问题尚未得到充分解答,目前只知道R2元件的这种靶向性需要3'UTR中的一个元素,但这个元素具体的位置还没有确定。3'UTR(3' untranslated region)指RNA分子的3'端非编码区。
为此,张锋团队使用冷冻电子显微镜解析了R2元件在28S rRNA基因上使用自身3' UTR启动TPRT的结构。该结构揭示了3' UTR中与目标DNA产生交互的核心区域,并表明,可以通过对R2元件进行改造,使其靶向28S rRNA基因以外的位点。
研究发现,R2Bm蛋白的核心是一个逆转录酶(RT)结构域,前后分别是一个特征性的N端扩展域和一个C端ɑ螺旋拇指结构域。R2Bm蛋白、3' UTR RNA和目标DNA之间存在几个关键的相互作用:目标DNA的两条链在ZnF域周围分离,其中底链(被切断的那条DNA链)进入限制性核酸内切酶(RLE)活性位点,而顶链沿着RLE的相反面蛇行;目标DNA与3' UTR RNA形成的异源双链被RT活性位点包含;3' UTR RNA经N端扩展域被引入到RT活性位点。
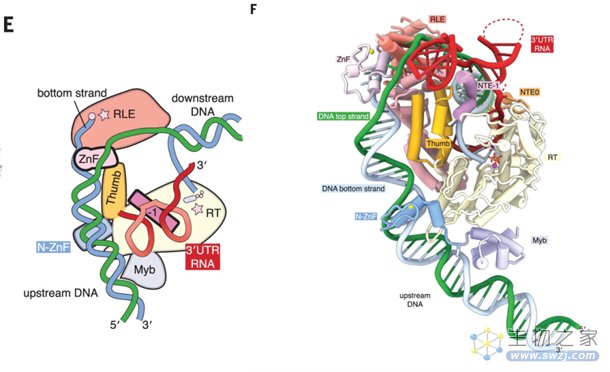
图4 R2Bm 反转录转座子的冷冻电镜结构(图源:[1])
研究团队还发现了目标28S DNA序列上与可能与R2Bm特异性识别有关的两个关键区域:其一是-34到-22的上游基序,与N端N-ZnF和Myb结构域结合;其二是-6到+1,与RLE结合。研究人员称之为逆转录转座子上游基序(Retrotransposon Upstream Motif,RUM)和逆转录转座子相关插入位点(Retrotransposon-Associated INsertion site,RASIN)。
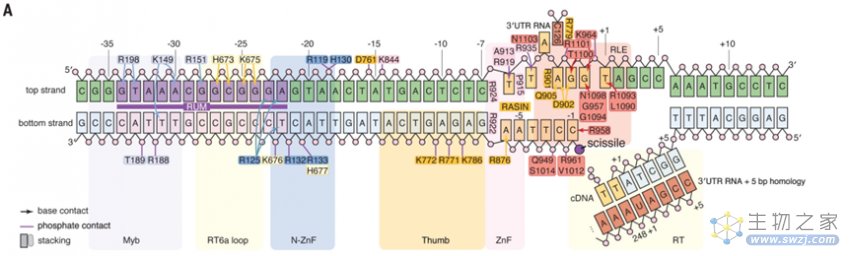
图5 R2Bm与目标DNA相互作用示意图(图源:[1])
研究团队推断,TPRT的启动包含以下步骤:R2Bm的N端结构域首先检测RUM序列,然后在 RASIN位点切割底链,可能将剪切位点绕顶链旋转到RT活性位点,将任何3'同源序列与剪开的底链配对后,最终启动逆转录。进一步的实验表明,R2Bm可以在外源性底链附近启动逆转录,并且能在Cas9的指导下在28S DNA序列以外的目标位点执行TPRT。
有意思的是,以上结果表明,不同于其他非LTR逆转录转座子,仅靠核酸内切酶结构域决定目标位点的选择,R2Bm使用其N-ZnF和Myb结构域来定位核酸内切酶的目标序列。此外,研究团队还发现,RUM-RASIN共识基序搜索家蚕基因组的结果提示,存在许多脱靶位点,但实际情况中,非28S插入非常少见,这可能是其他因素调节了R2Bm的转座。
总而言之,这项研究就非LTR逆转录转座子给出了新颖而深刻的理解。Cas9成功指导R2Bm重新定向更表明R2Bm未来有望作为一种新的基因插入工具发挥更大的作用。
参考资料:
[1]Wilkinson ME, Frangieh CJ, Macrae RK, et al. Structure of the R2 non-LTR retrotransposon initiating target-primed reverse transcription. Science. 2023 Apr 6:eadg7883. doi: 10.1126/science.adg7883.
[2]https://www.jove.com/science-education/11574/non-ltr-retrotransposons