1865年,奥地利小城布尔诺当地的自然研究学会上,一位微胖的传教士正在做一个长篇学术报告,报告的内容是他花了八年时间在圣托马斯修道院进行的豌豆杂交实验所得出的成果。
通过这些实验,他认为生物性状的遗传是受遗传因子控制的,并提出了遗传因子分离和自由组合的基本遗传规律。

图:《豌豆杂交实验》论文手稿
不过,他的发现如同小石子投进大海,在之后的数十年里没有泛起大的波澜。直到许多年后,这位传教士的名字格雷戈尔·孟德尔才以“现代遗传学之父”的称号的留名青史。
划时代的研究具有一个共性:不仅为迫在眉睫的问题给出答案,更为之后的研究打开新的天地。孟德尔的研究激励一代代人破解生物遗传密码:弄清楚“遗传因子”究竟是什么及其背后机制,成为生物学家们孜孜以求的科学高峰。
01
前基因时代:插上想象力的翅膀
1909年,丹麦遗传学家约翰逊(W.Johansen 1859~1927)首次提出了“基因”这一说法,以此来替代孟德尔假定的“遗传因子”,虽然此时的基因不过是一个没有任何实证的概念。
20世纪40年代,那个可能是有史以来最为知名的"猫奴"、量子力学奠基者薛定谔,将物理学引入生物学之中,在其所著的《生命是什么?》,他设想生命的遗传物质是一种“非周期性晶体”,而遗传变异则是“基因分子的量子跃迁”。
图:薛定谔
那时的人们虽然已经发现了核酸之中有四种常见碱基:腺嘌呤(A)、胸腺嘧啶(T)、胞嘧啶(C)和尿嘧啶(U),但对于这四种分子是以什么方式组合在一起等问题仍然莫衷一是。
1953年,沃森和克里克发现了DNA双螺旋的结构,“生命之谜”终于向勇于挑战的人掀开了她神秘的面纱,分子生物学时代就此开启,广阔的基因世界向人类敞开。

图:沃森(左)与克里克(右)
但必须说明的是,DNA双螺旋结构的提出并非严谨实验得出的结果,而是沃森和克里克将前人的成果(如富兰克林的X光衍射和查加夫规则)进行借鉴,再加上二人的天才构想进一步推理而来。
不得不感叹,在人类进步的道路上,想象力总是最伟大的先行者。
02
基因工程与数据爆炸
认识到DNA的结构只是万里长征的第一步,如何进一步干预和操控基因成为此后的数十年生物领域的热门议题,使得数千名科学家投身其中。
基因工程,顾名思义,就是将基因作为工程化的对象,使之按照人们的意愿遗传并表达出新的性状。
20世纪60年代初,科学家发现了限制性内切酶和DNA连接酶等,实现了DNA分子体外切割和连接。1972年保罗·伯格首次构建了重组DNA分子,提出了体外重组DNA分子是如何进入宿主细胞,并在其中复制和有效表达等问题。至此基因重组的基本技术几乎同时于20世纪70年代初得以建立,基因工程的诞生只差临门一脚。
1973年,美国斯坦福大学的科恩(Cohen)和博耶(Boyer)等人在体外构建出含有四环素和链霉素两个抗性基因的重组质粒分子,将之导入大肠杆菌后,该重组质粒得以稳定复制,并赋予受体细胞相应的抗生素抗性,由此宣告了基因工程的诞生。

图:强者发型的科恩
此后基因工程不断发展,并发展出基因敲入、基因敲除、基因表达调控和基因编辑四大应用方向。
但此时的“基因”,对人类来说依旧是一个相当模糊的概念,直到1977年,弗雷德里克·桑格突破性的使用双脱氧链终止法完成了世界上第一次核酸测序,曾经神秘莫测的“基因”变成了一系列可读的核苷酸序列。
生物的基因序列蕴藏着全部遗传信息,准确地确定序列在基因的分离、定位、结构与功能的研究,基因工程中载体的组建,基因表达与调控基因片段的合成和探针的制备,基因与疾病的关系等方面都有十分重要的作用。
发端于上世纪80年代的基因组学(“组”在基因组一词中,意指一个物种的全部遗传组成。)在过去的数十年内快速发展,2000年首次商用的高通量测序(High-throughput Sequencing, THS)让大规模、低成本、快速地获得基因序列成为可能。
预计到 2025 年,所有人类基因组数据将达到40 EB(1EB=2^30GB)。作为参考,这比存储历史上所说的每个单词所需的存储空间多 8 倍。
03
深度学习:基因领域大杀器
如此庞大的数据,如何分析和处理就成了难题。
好在AI技术的出现为科学家们提供了一种高效的解决方案,帮助将异质性和大规模的基因组数据集转化为生物学上的结论,其中尤以深度学习(Deep Learning,又被称为深度神经网络)为最。
深度学习的基本架构发端于人类大脑中的生物神经网络,通常由三个层组成:输入层、隐藏层和输出层。数据从输入层输入,通过一个或多个隐藏层的加权,最终结果将在输出层呈现出来。
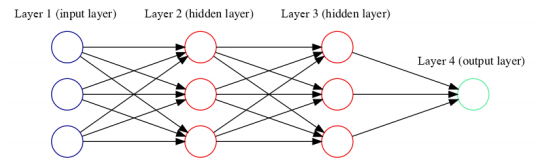
图:有两个隐藏层的深度学习模型
其中层次结构中的每个算法(图中的圆,即神经元)都使用一个非线性变换函数作为输入来预测输出。不需要人工干预的迭代学习会不断进行,直到预测的输出达到期望水平。
深度学习对于那些收集、分析和解释大量数据的学科非常有用,其不仅提供了一种分析某一特定类型数据的强大方法,而且还能将多种互补方法的数据结合起来并找出数据中的各种隐含关系,因此非常适合基因组学。
目前常用的几种深度学习框架包括前馈神经网络(FNN)、卷积神经网络(CNN)、循环神经网络(RNN)、图神经网络(GNN)、生成对抗网络(GAN)等,而目前在基因组学中最为常用的是卷积神经网络。
在卷积神经网络产生之前,识别图像中的对象需要手动的特征提取方法。但是,当目标操作更为复杂的时候,由于参数过多,单靠手工难以实现。
卷积神经网络是一种基于卷积计算的前馈神经网络,由卷积层、池化层和全连接层组成。其中,卷积层负责提取图像中的局部特征;池化层用来大幅降低参数量级;全连接层类似传统神经网络的部分,用来输出想要的结果。
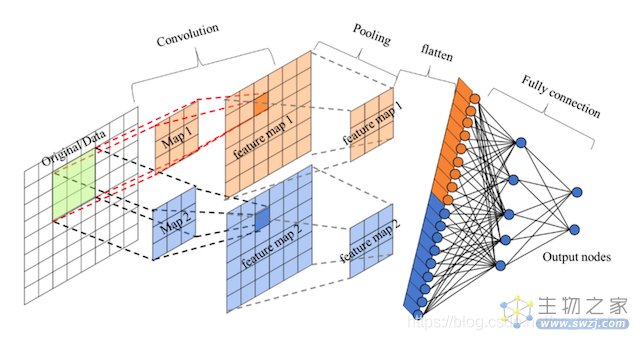
图:卷积神经网络模型
与普通的神经网络相比,卷积神经网络具有参数量低、自动提取特征等优势,被广泛应用于图像识别、图像分类、目标检测、轮廓识别等问题领域。
仅在基因编辑领域,就已有多个基于CNN的模型:
(1)DeepCas9:直接从sgRNA序列特征中学习并预测具有高活性的sgRNA(sgRNA是CRISPR/Cas9系统的重要组成部分,用于识别特异的DNA序列),有助于减少必须通过实验验证的sgRNA的数量,以实现更有效和高效的遗传筛选和基因组工程。
(2)DeepCRISPR:将中靶位点与脱靶位点的预测整合都一个深度学习框架中以优化gRNA的设计,DeepCRISPR从人类编码和非编码区域提取大约5亿个未标记的gRNA序列,并学习gRNA的低维特征,其还使用了一种数据增强方法,创建了不到一百万个的具有已知敲除效率的sgRNA来训练一个更大更复杂的CNN模型,以达到更好的预测效果
(3)ADAPT:构建了一个大规模的CRISPR训练数据集,可用于脊椎动物感染病毒诊断的全自动CRISPR设计。
04
深度学习在基因组学的广泛应用
越来越多的深度学习已经深入到基因组学的各个方向,以下是一些典型:
基因表达预测
基因表达是指基因经转录、翻译形成有生物活性RNA或蛋白质的过程,对于生物维持生命活动和适应外界环境具有关键作用。
人类基因组的大约 30 亿对DNA 碱基中,只有约2%对应于编码蛋白质的基因,剩下的98%都属于非编码DNA,科学家对此了解甚少。
进一步了解基因表达的原理并进行预测,对于人类探索自身意义重大。
2021年10月,DeepMind携手Calico在国际知名学术期刊 Nature Methods上在线发表题为 Effective gene expression prediction from sequence by integrating long-range interactions 的文章,引入了一种叫做 Enformer 的神经网络架构,大大提高了根据 DNA 序列预测基因表达的准确性。为了进一步研究疾病中的基因调控和致病因素,研究者还公开了他们的模型及其对常见遗传变异的初步预测。
基因表达调控
在严格的调控系统下,生物体内的基因表达在时间和空间上呈现有序性,所以人们为了改变生物体某种目的产物的水平,需要利用基因调控手段来实现,包括不同类型启动子原件的应用、翻译起始区mRNA的优化、密码子的优化以及转录因子的应用。
其中转录因子(TF)是指一类能够结合在特异的DNA序列上来抑制或者激活特定基因转录的调控蛋白。转录因子能够以特定序列与基因专一性结合,保证目的基因以特定的强度在特定的时间空间表达。
去年4月,中国军事科学院军事医学研究院伯晓晨团队在人工智能顶级期刊Nature Machine Intelligence上发表了题为《Inferring transcription factor regulatory networks from single-cell ATAC-seq data based on graph neural networks 》的研究论文
研究采用图神经网络技术,提出了一种基于染色质结构的转录因子调控网络建模方法DeepTFni,可以帮助生物医学研究人员透过纷繁复杂的组学观测数据锁定关键的功能分子和通路,从而克服人工推理的局限性,显著提高研究效率。
单细胞测序
单细胞测序技术是指在单个细胞水平上,对基因组等遗传信息进行高通量测序分析的一项技术。
与传统测序相比,单细胞测序能够在更精细的单个细胞水平上获得遗传信息,从而揭示每个细胞的基因结构和基因表达状态,反映异质性信息和稀有信息。
去年腾讯AI Lab 团队在国际顶级学术期刊《Nature》子刊《Nature Machine Intelligence》发表文章,提出了一种用于单细胞RNA-seq数据的细胞类型注释的大规模预训练语言模型scBERT,首次将Transformer架构运用到单细胞转录组测序数据分析领域。
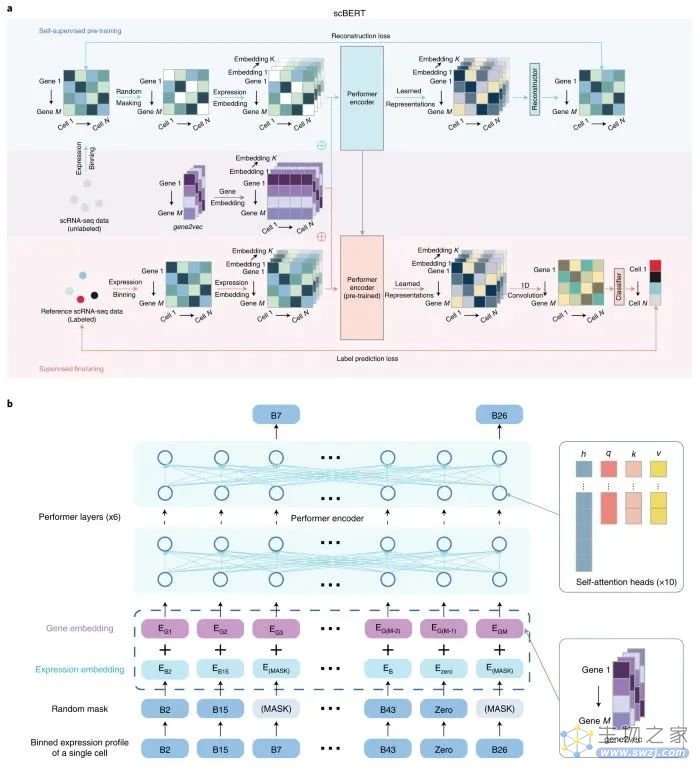
图:scBERT模型
遵循 BERT 的预训练和微调方法,scBERT 通过对大量未标记的 scRNA-seq 数据进行预训练,对基因-基因相互作用有了大致的了解;然后将其转移到未见和用户特定的 scRNA-seq 数据的细胞类型注释任务,以进行监督微调。
研究验证了 scBERT 在细胞类型注释、新细胞类型发现、批处理效应的稳健性和模型可解释性方面的卓越性能。
写在最后
上世纪40年代,生物学尚处于萌芽阶段,人们对其生命背后的机制知之甚少。
不过,正如人类在物理、化学所取得的成就一样,科学家们相信将生命当作一种研究客体,总能发现其所遵循的法则。
薛定谔说:“在有机体的生命周期里展开的事件中,显示出一种美妙的规律性和秩序性,我们碰到过的任何一种无生命物质都是无法与之相比的。”
然而,这位伟大的物理学家并没有仅仅将生物学机械地与其他学科化为等号。
他明白无论生命再怎么作为客体对象被对待,由于人的参与(人本身就是生命的一种形式),其终究保留着不可理解的那一层面。“根据已知的生命物质结构,我们一定会发现,它的工作方式是无法归结为物理学的普通定律的”。
这超过了大多数生物科学研究力所能及的范围,传统的还原论方法几乎没办法开展对这些问题的处理。
好在近年来人工智能尤其是深度学习技术的突飞猛进,为我们提供了一种易于处理的分析方法来替代:从整体出发,尽可能地收集数据,让AI从生物系统中找出输入和输出之间的复杂关系并构建捕获潜在规则的模型。
当然,新技术的使用会使人们不可避免地陷入焦虑,毕竟面对深度学习的“黑箱”,我们并不知道它们是怎么做到的。
在这一过程中,诚实就显得无比珍贵:承认自身的局限性,并付出严肃认真的努力,踏入从未踏入的领域,寻找原先深信无法找到的事物。
最终,在了解并接纳生命的复杂性后,AI将成为我们的利器。