大约 7 年前,美国艾伦细胞科学研究所(Allen Institute for Cell Science)刚成立不久。那时,该研究所试图找出用荧光蛋白标记细胞内结构的最佳方法,以便既能 " 看到 " 它们,同时又尽可能地维持细胞的正常状态。
通过上述努力,该团队创建了人类诱导多能干细胞系的 " 艾伦细胞集 "(Allen Cell Collection)。在这之后,他们尝试对这些活细胞进行三维成像,以便能以足够高的分辨率看清楚细胞的结构。这样一来,就能在不杀死细胞的情况下,对所有细胞进行分析。
不过,还得先完成一个非常艰巨的任务,就是开发图像分析工具,将原始三维图像转换成单个细胞及其内部结构的图像。
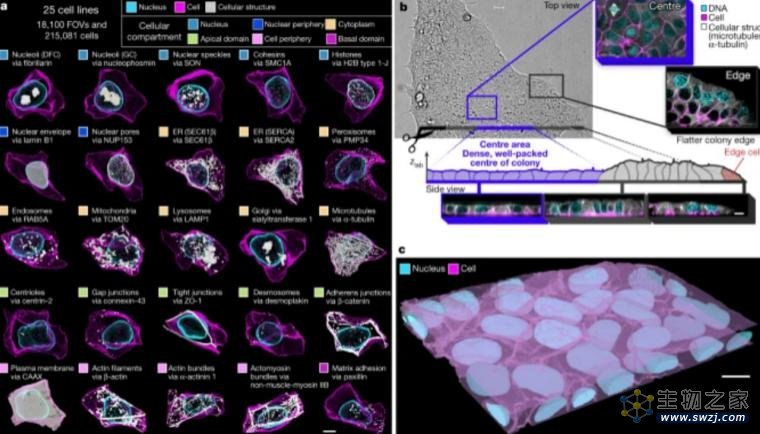
完成上述任务之后,一个数据集也随之诞生。这让研究人员可以开发通用的分析框架。该框架基于两个坐标系统:一个用于细胞形状,一个用于细胞内部结构的映射。
(来源:Nature)
最近,该团队在 Nature 发表了一篇题为《整合细胞内组织及其在人 iPS 细胞中的变异》()的论文,共有 84 位作者。

图 | 相关论文(来源:Nature)
曾就职于艾伦细胞研究所(目前在德国莱布尼茨分析科学研究所担任研究组组长)的陈建旭,也参与了这项研究。
在他的引荐之下,艾伦细胞研究所科研副主任苏珊娜 · 拉菲尔斯基(Susanne Rafelski)告诉 DeepTech:" 论文包含了该研究所各个分支团队的几乎所有专业力量,是真正的共同努力的结果。专家们的领域涵盖分子生物学、细胞生物学、计算科学、软件工程、工具可视化等。"

图 | Susanne Rafelski(来源:资料图)
研究中,课题组揭示了正常细胞用于组织自身的基本规则,借此可以了解一个细胞到底是什么样子,它的内部组件是如何布局的,以及如何从根本上与细胞的行为方式联系在一起。
当然,不同细胞之间也会存在一些 " 正常 " 的差异。那么,如何判断在一个细胞中看到的差异,到底是正常变化的一部分、还是疾病的预警?可以说,此次论文的面世,让研究人员拥有了一种可以严格、定量地确定细胞差异的方法。
以这种方法所获的数据为参考,就能对健康和疾病的细胞基础,从本质上产生更深刻的了解。当我们知道一个 " 正常 " 细胞群所有可能的存在方式时,就可以更准确地确定当细胞群出现异常时,到底问题出现在哪里,从而开发更有效的疾病治疗方法。
此次工作的出色之处在于,研究人员用来分析和可视化特定细胞的方法,也可以用于身体中的许多其他细胞,就像一本指南一样。
例如,研究人员已将这个指南中的一些概念,用于分析其他细胞类型和状态下的细胞组织,除了研究细胞内部的结构之外,他们还在寻找构成这些结构的蛋白质在位置组成上的模式。
Susanne Rafelski 说:" 当我第一次看到将细胞结构的平均位置,被绘制成干细胞的平均形状的方法时,这与我多年来仔细观察的数千张干细胞个体图像完美匹配到一起。并准确地总结出所有结果的意义时,我知道这种计算方法在生物学上是有效的,因此,我们现在可以应用它来提出基本的生物学问题。"
而此次论文的发表,也意味着相关的框架已经建立。" 秉承着开放科学的价值观,我们希望世界各地的科学家可以使用本次方法来研究他们自己的细胞系,并发现细胞行为和组织的基本原则。" 研究人员表示。
据介绍,揭示 " 正常细胞 " 的规则、以及了解 " 正常细胞 " 所有可能的变化,是帮助人们找到更好的疾病治疗方法的关键。该团队已将论文中的一些概念用于分析其他细胞类型和状态的细胞组织,并将这些概念用于寻找蛋白质的位置模式。
参考资料:
1.Viana, M.P., Chen, J., Knijnenburg, T.A. et al. Integrated intracellular organization and its variations in human iPS cells. Nature 613, 345 – 354 ( 2023 ) . https://www.nature.com/articles/s41586-022-05563-7
https://cfe.allencell.org
https://alleninstitute.org/what-we-do/cell-science/news-press/articles/interior-design-our-cells